Chi era il il Prof. Guido Tarone

Nato a Torino nel 1951, il Prof. Guido Tarone si è laureato in Scienze Biologiche all’Università di Torino nel 1974 con la votazione di 110/110 e Lode. Il Prof. Guido Tarone è prematuramente scomparso nel maggio del 2015, lasciando un grande vuoto nella sua famiglia e tra gli amici e i colleghi.
Guido Tarone è stato uno scienziato di fama internazionale nel campo della Biologia Cellulare. Il suo interesse scientifico si è rivolto alla comprensione dei meccanismi che permettono alle cellule di comunicare tra loro e con l’ambiente circostante. Nel corso della sua carriera Guido Tarone ha contribuito all’identificazione di un’importante classe di proteine cellulari, le integrine, fondamentali per la comunicazione tra le cellule e la matrice extracellulare, coinvolte in processi fisiologici di base, quali la differenziazione delle cellule e la loro proliferazione e deregolate in moltissime patologie.
I suoi studi hanno permesso di identificare e caratterizzare nuove isoforme delle integrine coinvolte nell’adesione e nella regolazione della contrattilità della cellula muscolare cardiaca. Negli ultimi 10 anni della sua carriera ha lavorato ai meccanismi di segnalazione cardiaca identificando meccanismi per controllare il programma di ipertrofia compensatoria, lavorando, grazie anche a fondi Telethon, allo sviluppo di un protocollo di terapia genica per contrastare le cardiopatie genetiche.
I risultati del lavoro sperimentale sono riportati in 137 pubblicazioni a stampa su riviste internazionali e numerose comunicazioni a congressi.
Il Prof. Tarone ha svolto la sua attività in prestigiosi Centri di ricerca all’estero, in USA, tra cui il Dipartimento di Patologia dell’Università di Yale, il Winstar Institute di Filadelfia, il La Jolla Cancer Research Center e il Dipartimento di Patologia della Columbia University. Rientrato in Italia, ha lavorato all’Università di Trieste e alla Sapienza di Roma, mantenendo sempre l’Università degli Studi di Torino come punto di riferimento. In questa Università, oltre che alla ricerca, si è dedicato alla formazione dei giovani, dirigendo la Scuola di Dottorato e contribuendo attivamente alla costruzione del Corso di Biotecnologie. La didattica lo ha visto sempre profondamente coinvolto perché convinto che solo attraverso l’educazione dei giovani si può ottenere il progresso economico e civile.
Inoltre, il Prof. Tarone ha diretto il Dipartimento di Biotecnologie Molecolari e Scienze per la Salute a cui molto si è dedicato sia nella fase costitutiva, con l’aggregazione di gruppi di ricerca sinergici, che nella fase realizzativa preparando per tutti le migliori condizioni di lavoro.
È stato responsabile scientifico di innumerevoli progetti di ricerca finanziati dal MUR, AIRC, Telethon, EU e coordinatore nazionale di progetti PRIN, FIRB e Progetti Regionali. Ha ricoperto numerose cariche in diverse società scientifiche: è stato Presidente della Società Italiana Biologia Cellulare e del Differenziamento (ABCD) dal 2005 al 2008; dal 2012 Vice presidente del Gruppo di Lavoro “Myocardial function” della Società Europea di Cardiologia di cui, dal 2014, è diventato Presidente. È stato membro di Comitati Internazionali per la Valutazione delle attività scientifica nei centri di ricerca Biocenter Oulu, University of Oulu e Institut de Biologie et Chimie des Protéines, Lyon.
Dal 2010 al 2012 è stato membro del Comitato Tecnico Scientifico dell’Associazione per la Ricerca sul Cancro (AIRC).
Al rigore scientifico Il Prof. Guido Tarone ha sempre affiancato grande umanità ed entusiasmo, nonché una grande disponibilità a mettersi a disposizione dei ricercatori più giovani ed è stato un esempio per moltissimi ricercatori che hanno avuto l’onore di incontrarlo e lavorare con lui.
Vincitori del Premio Tarone per Giovani Ricercatori
XXI Congresso Nazionale di Salerno – 2024

Caterina Bernacchioni – Under 45
Department of Experimental and Clinical Biomedical Sciences “Mario Serio” (SBSC) – University of Florence

Roberto Oleari – Under 35
Department of Pharmacological and Biomolecular Sciences – University of Milan
SEMA6A drives GnRH neuron-dependent puberty onset by tuning median eminence vascular permeability.
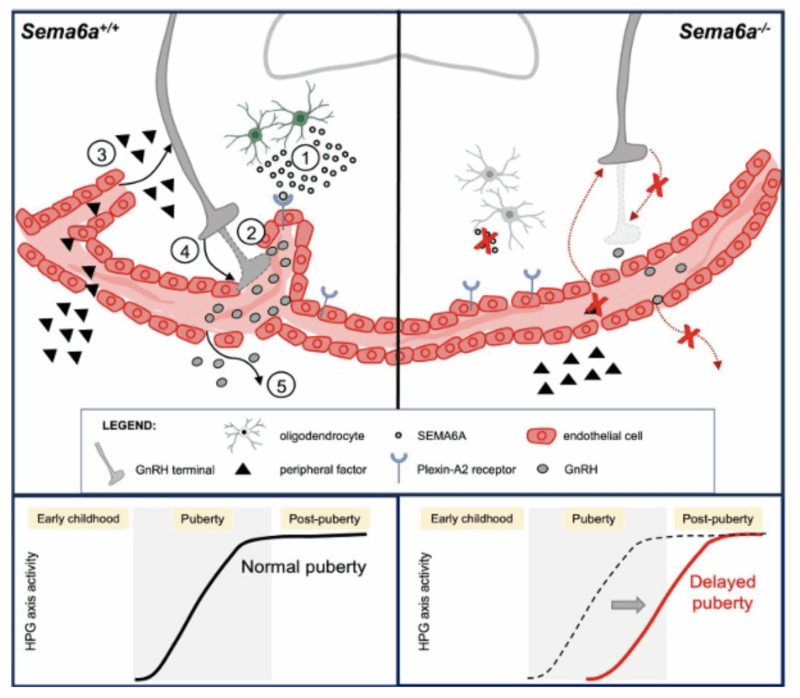
Gonadotropin-Releasing Hormone (GnRH) neurons control reproduction in most vertebrates and, during embryogenesis, migrate from the nose to the hypothalamus. There, the innervation of the median eminence (ME) by GnRH+ axons is vital to ensure puberty onset. However, molecular and cellular mechanisms underlying ME development and genetic determinants of pubertal timing are incompletely understood. Here, we found a novel SEMA6A variant in a 6 proband from 3 unrelated families presenting delayed puberty. First, we demonstrated that the identified variant is pathogenic by in silico analyses and affects protein synthesis and localization upon overexpression in COS-7 cells. Then, by analyzing wildtype and Sema6a-null embryos, we showed that SEMA6A is expressed during GnRH neuron embryonic development, but is dispensable for their migration. Yet, SEMA6A expression in the ME raised the possibility of SEMA6A additional roles on GnRH neuron within the postnatal brain. In agreement with this hypothesis, we found that SEMA6A is expressed by ME-resident oligodendrocytes (1) and its loss significantly impairs GnRH axon innervation of the ME (2); consequently, puberty onset was significantly delayed in male and female Sema6a-null mice. Finally, we investigated cellular and molecular mechanisms through which SEMA6A control these processes. First, we found that SEMA6A receptor PLXNA2 is expressed by fenestrated endothelial cells lining the ME, suggesting a possible involvement of SEMA6A in regulating neurovascular interactions. Then, we demonstrated that SEMA6A is able to enhance the permeability of endothelial cells in a 2D in vitro assay by coupling with PLXNA2 receptor. Further, we confirmed that SEMA6A loss in vivo reduces vascular permeability and the number of fenestrated loops in the ME. In all, our data revealed a hitherto undescribed role for SEMA6A in tuning the ME vascular barrier and for proper puberty onset, possibly modulating the exchange of molecules (3) key for GnRH neuron patterning (4-5).
XXI Congresso Nazionale di Bari – 2023

Rosanna Chianese – Under 45
Department of Experimental Medicine – University of Campania “L. Vanvitelli”

Barbara Tedesco – Under 35
Department of Pharmacological and Biomolecular Sciences (DiSFeB) “Rodolfo Paoletti”
FRAMESHIFT MUTATIONS IN THE HEAT SHOCK PROTEIN B8 CAUSE ITS AGGREGATION AND IMPAIR PROTEOSTASIS IN NEUROMYOPATHIES
The heat shock protein B8 (HSPB8) is a chaperone highly expressed in muscle cells, where it favors the chaperone-assisted selective autophagy (CASA) of damaged structural proteins. In neurons, HSPB8 enhances the clearance of misfolded proteins associated with motoneuron diseases. This is achieved by the interaction of HSPB8 with the cochaperone BAG3, forming the CASA complex together with the heat shock protein family A (HSPA) and the E3-ubiquitin ligase STUB1. The chaperones of the CASA complex recognize the substrate, which might undergo refolding or STUB1-mediated ubiquitination. Therefore, ubiquitinated proteins can be targeted to autophagic degradation. Different frameshift mutations in the HSPB8 gene have been identified in neuromyopathies. These HSPB8 frameshift mutations cause the elongation of the HSPB8 protein product at the carboxy-terminus and a variable modification of the carboxy-terminal domain. Here, we show that the HSPB8 frameshift mutants are characterized by high insolubility and aggregation propensity. The HSPB8 frameshift mutants retain the ability to take part in the CASA complex, determining the sequestration of the HSPB8 wild-type and the other components of the CASA complex. As a result, misfolded and ubiquitinated substrates are entrapped in HSPB8-mutant aggregates together with autophagy receptors. Notably, we show that HSPB8 mutant aggregation is driven by neither the other CASA members nor autophagy receptors. Instead, we found that the mutated carboxy-terminal sequence of the HSPB8 mutants possesses intrinsic properties for aggregation. In summary, here we describe a gain of toxic function mechanism through which different HSPB8 frameshift mutations may cause neuromuscular diseases
XX Congresso Nazionale di Roma – 2022

Manuela Basso – Under 45 (ex aequo)
Department of Cellular, Computational and Integrative Biology – CIBIO – University of Trento

Anna Maria Cariboni – Under 45 (ex aequo)
Department of Pharmacological and Biomolecular Sciences – University of Milan

Federico Gulluni – Under 35
Department of Molecular Biotechnology and Health Sciences Molecular Biotechnology Center “Guido Tarone” – University of Turin
Aneuploidy, characterized by an abnormal number of chromosomes, is a well-established characteristic linked to cancer development. Unlike normal cells, which usually maintain a stable and predictable number of chromosomes (euploid), cancer cells frequently exhibit an erratic count (aneuploid).
In our research focused on breast cancer, we explore the deficiency of phosphoinositide kinases. When these kinases are dysfunctional, they disrupt the normal arrangement and segregation of chromosomes during cell division, resulting in genomic instability.
The objective of our research is to comprehend the significance of aneuploidy in cancer and to determine whether it presents vulnerabilities that can be targeted for treatment.
XVIII Congresso Nazionale di Ferrara – 2018
Cristina Barbagallo – Under 35
Department of Biomedical and Biotechnological Sciences – University of Catania
LncRNA UCA1, Upregulated in CRC Biopsies and Downregulated in Serum Exosomes, Controls mRNA Expression by RNA-RNA Interactions
Long non-coding RNAs (lncRNAs) and circular RNAs (circRNAs) contribute to the onset of many neoplasias through RNA-RNA competitive interactions; in addition, they could be secreted by cancer cells into biological fluids, suggesting their potential diagnostic application. By analyzing the expression of 17 lncRNAs and 31 circRNAs in biopsies and serum exosomes from colorectal cancer (CRC) patients through qRT-PCR, we detected CCAT1, CCAT2, HOTAIR, and UCA1 upregulation and CDR1AS, MALAT1, and TUG1 downregulation in biopsies. In serum exosomes, UCA1 was downregulated, while circHIPK3 and TUG1 were upregulated. Combined receiver operating characteristic (ROC) curves of TUG1:UCA1 and circHIPK3:UCA1 showed high values of sensitivity and specificity. Through in vitro (i.e., RNA silencing and mitogen-activated protein kinase [MAPK] inhibition) and in silico analyses (i.e., expression correlation and RNA-RNA-binding prediction), we found that UCA1 could (1) be controlled by MAPKs through CEBPB; (2) sequester miR-135a,miR-143,miR-214, andmiR-1271, protecting ANLN, BIRC5, IPO7, KIF2A, and KIF23 from microRNA (miRNA)- induced degradation; and (3) interact with mRNA 30-UTRs, preventing miRNA binding. UCA1 and its co-regulated antisense LINC01764 could interact and reciprocally mask their own miRNA-binding sites.
Functional enrichment analysis of the RNA-RNA network controlled by UCA1 suggested its potential involvement in cellular migration. The UCA1 regulatory axis would represent a promising target to develop innovative RNA-based therapeutics against CRC.
Citation: Barbagallo C, Brex D, Caponnetto A, Cirnigliaro M, Scalia M, Magnano A, Caltabiano R, Barbagallo D, Biondi A, Cappellani A, Basile F, Di Pietro C, Purrello M, Ragusa M. LncRNA UCA1, Upregulated in CRC Biopsies and Downregulated in Serum Exosomes, Controls mRNA Expression by RNA-RNA Interactions. Mol Ther Nucleic Acids. 2018 Sep 7;12:229-241. doi: 10.1016/j.omtn.2018.05.009. Epub 2018 Jun 2. PMID: 30195762


